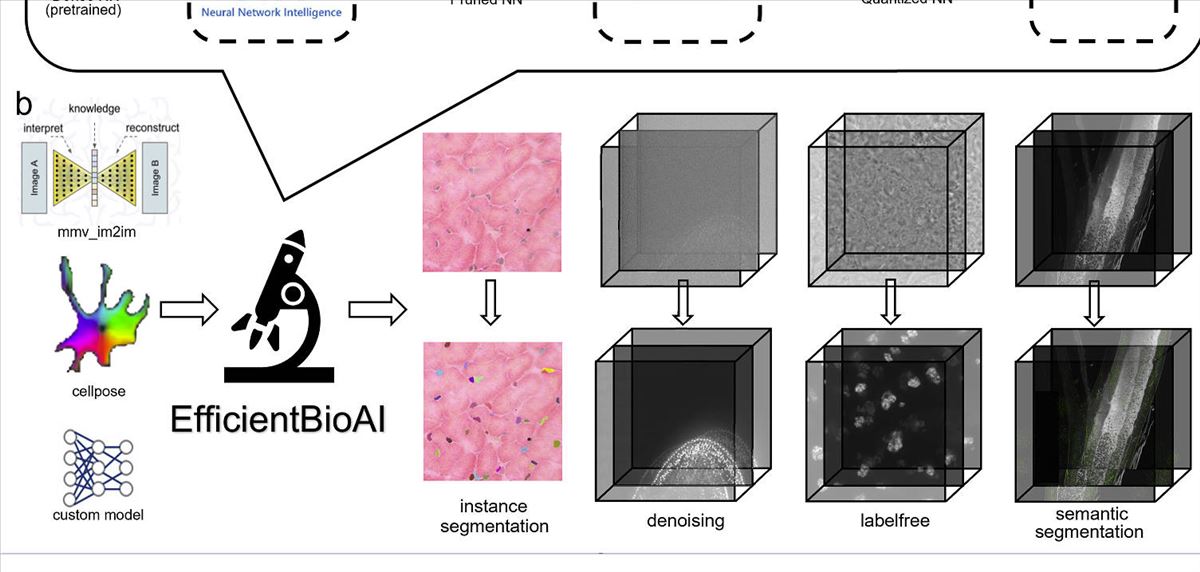
La inteligencia artificial (IA) ha desempeñado un papel fundamental en la revolución de la tecnología de la microscopía y el análisis de datos microscópicos. Sin embargo, a medida que los modelos de IA se vuelven más complejos, su demanda de potencia de cálculo y consumo de energía también aumenta. En este contexto, un equipo de investigadores del Leibniz-Institut für Analytische Wissenschaften (ISAS) y la Universidad de Pekín ha desarrollado un software de compresión de código abierto llamado EfficientBioAI, disponible en GitHub, con el objetivo de permitir que los científicos ejecuten modelos de IA en bioimágenes de manera más eficiente, reduciendo tanto el tiempo de procesamiento como el consumo de energía.
El desafío de la eficiencia en bioimágenes
En la actualidad, las técnicas modernas de microscopía generan una gran cantidad de imágenes de alta resolución, lo que da como resultado conjuntos de datos que pueden constar de miles de imágenes. Los científicos confían en software respaldado por IA para analizar estos conjuntos de datos de manera confiable. Sin embargo, a medida que los modelos de IA se vuelven más intrincados, el tiempo de procesamiento de las imágenes aumenta, lo que conduce a una mayor demanda de potencia de cálculo y consumo de energía.
Según el Dr. Jianxu Chen, jefe del grupo de investigación junior AMBIOM (Análisis de Imágenes BIOMédicas Microscópicas) en el ISAS, la alta latencia de la red, especialmente con imágenes grandes, contribuye a estos desafíos. Para mitigar estos problemas, los investigadores han recurrido a técnicas de compresión de modelos, ampliamente utilizadas en visión por computadora y IA, para hacer que los modelos de IA sean más ligeros y eficientes en términos de energía.
EfficientBioAI: Una solución para la compresión de modelos de IA
EfficientBioAI utiliza diversas estrategias para reducir el consumo de memoria y acelerar la inferencia del modelo. Una de estas estrategias es la poda, que implica la eliminación de nodos innecesarios de la red neuronal. Este enfoque permite a los científicos optimizar la eficiencia de las redes neuronales en bioimágenes sin comprometer la precisión del modelo.
Para evaluar la efectividad de EfficientBioAI, los investigadores llevaron a cabo pruebas en aplicaciones de la vida real, utilizando diferentes configuraciones de hardware y diversas tareas de análisis de bioimágenes. Los resultados fueron prometedores, con las técnicas de compresión logrando reducciones sustanciales en la latencia y el consumo de energía, que oscilaron entre un 12.5% y un impresionante 80.6%.
Beneficios tangibles: ahorro de energía significativo
Para ilustrar el ahorro de energía logrado, el Dr. Chen utiliza el modelo CellPose como ejemplo. Si mil usuarios utilizasen EfficientBioAI para comprimir el modelo y aplicarlo al conjunto de datos Jump Target ORF (que consta de aproximadamente un millón de imágenes microscópicas de células), el ahorro de energía sería equivalente a las emisiones de un viaje en automóvil de aproximadamente 11,750 kilómetros.
Accesibilidad y futuro de EfficientBioAI
Uno de los aspectos más destacados de EfficientBioAI es su accesibilidad. Los autores tienen como objetivo ponerlo a disposición del mayor número posible de científicos en investigación biomédica. Los investigadores pueden instalar fácilmente el software e integrarlo en las bibliotecas existentes de PyTorch. Además, para modelos ampliamente utilizados, como Cellpose, los científicos pueden utilizar el software sin necesidad de realizar cambios en el código. El grupo de investigación también proporciona demos y tutoriales para respaldar solicitudes de personalización específicas, lo que facilita la adaptación del software a modelos de IA personalizados con ajustes mínimos en el código.
EfficientBioAI es un software de compresión de código abierto diseñado para modelos de IA en el campo de la bioimagen. Aunque es fácil de usar para aplicaciones estándar, también ofrece funciones personalizables, como niveles de compresión ajustables y la capacidad de cambiar entre la unidad central de procesamiento (CPU) y la unidad de procesamiento gráfico (GPU). Los investigadores están trabajando activamente en ampliar la compatibilidad de la herramienta, con el objetivo de hacerla disponible para MacOS, además de Linux (Ubuntu 20.04, Debian 10) y Windows 10.